
基于耐消化肽的核桃主要过敏原Jug r 2 的线性表位筛选
郝梦真, 李欣芮, 牟瑶, 陈锡, 车会莲*
(中国农业大学食品科学与营养工程学院 食品质量与安全北京实验室 北京 100083)
基金项目:国家自然科学基金项目(81573158)
作者简介:郝梦真(1998—),女,硕士生
通信作者:车会莲 E-mai: chehuilian@cau.edu.cn
研究背景与目的:
食物过敏原线性表位是过敏原结构及其致敏性关系研究的重要方面。目前,免疫信息学方法是一种预测过敏原线性表位的常见高效方法。考虑到大多数食物过敏是发生在胃肠道的免疫反应,食物过敏原在胃肠道内的消化情况以及消化产物是影响过敏原致敏性潜能的关键因素。树坚果尤其是核桃,是食物过敏导致致命或接近致命反应的主要原因,其中,Jug r 2是核桃中的主要过敏原。本研究将核桃过敏原体外胃肠液模拟消化降解产物与已知的线性表位和通过生物信息学预测得到的线性表位进行比较,并将抗消化肽段定位至模拟Jug r 2三维结构上,进一步认识Jug r 2的结构特点,为评估核桃过敏原Jug r 2免疫原性和免疫反应性,以及进一步研究导致食物过敏的过敏原分子结构条件提供试验参考。
研究方法:利用体外模拟胃肠消化获得分布于Jug r 2的耐消化肽,并利用HLPC-MS/MS对抗消化肽段的一级序列进行分析。同时,利用DNAStar Protean和ABCpred两种免疫信息学工具对Jug r 2的潜在线性表位进行预测。最后,比较Jug r 2上的耐胃肠道消化肽段与预测得到和文献报道已知的线性表位序列的重合性,并分析抗消化肽段在Jug r 2空间结构上的分布,以建立一套研究食物过敏原线性表位的新流程。
试验结果:利用DNAStar Protean系统预测Jug r 2,获得22条潜在B细胞线性表位。利用ABCpred在线工具预测Jug r 2获得14条B细胞线性表位。两种工具所预测的B 细胞线性表位存在很多重叠的部分,以两种工具预测的线性表位作为最终结果,不考虑存在争议的结果。最终通过两种生物信息学预测得到8条B细胞线性表位。
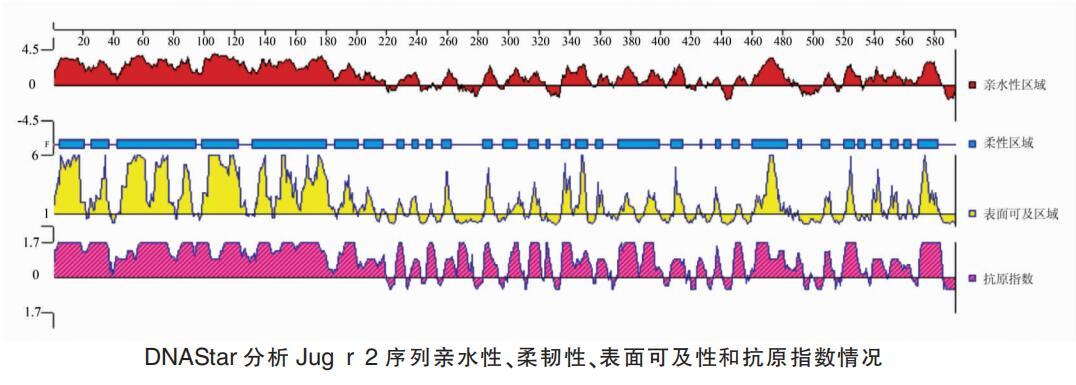
利用体外模拟胃液或模拟肠液对Jug r 2进行60 min的消化,经过根据Uniprot数据库提供的核桃过敏原氨基酸序列构建检索数据库,使用pFind软件搜索,分别鉴定出来源于Jug r 2的4条抗胃液消化肽段和13条抗胰液消化肽段。最终筛选得到11条的非重叠抗消化肽段。
Jug r 2的抗消化肽段在蛋白质全序列的AA545~572位置附近含有已知的B细胞线性表位,在AA250~260、AA363~388和AA470~483的抗消化肽与预测线性表位存在部分氨基酸序列的交叉,说明B细胞线性表位可能具有一定的抗消化性。除此之外,在AA215~220、AA250~260、AA323~337、AA363~388、AA428~438、AA470~483、AA545~572位置的抗消化肽段与已知的T细胞表位交叉。
将11条抗消化肽段定位于Jug r 2的三维模型结构中,以观察抗消化肽段的空间分布特点。本研究发现5条抗消化肽段位于转角结构,2条无规卷曲处。更多抗消化肽段位于Jug r 2的表面,亲水性良好,且这些肽段所处转角和无规卷曲结构具有可变性,会提高抗消化肽与IgE的结合活性。
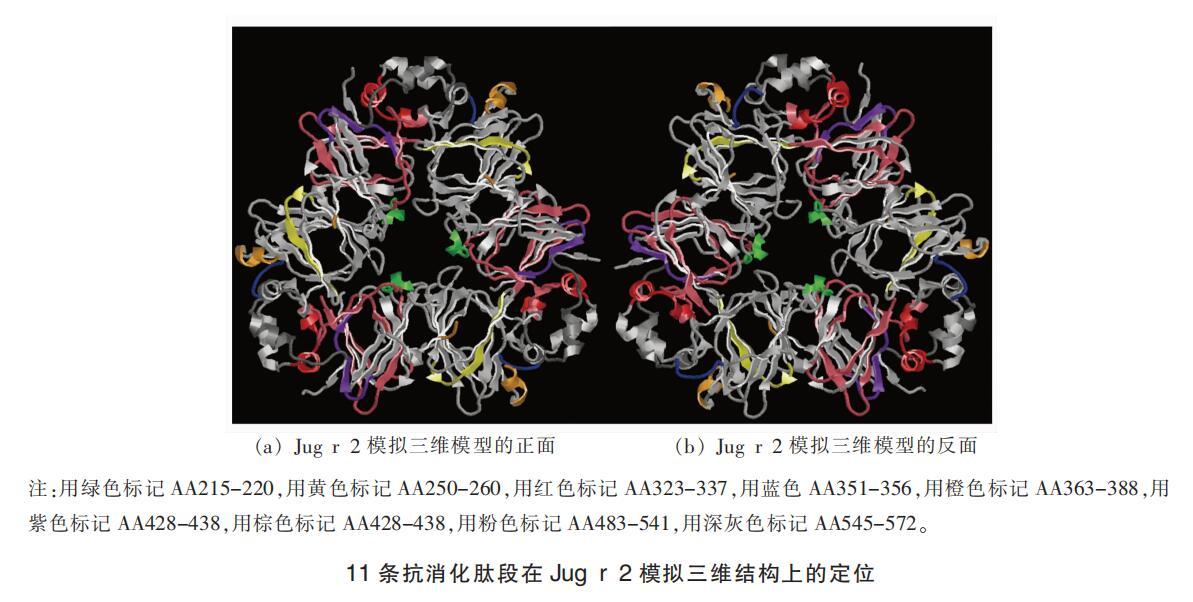
结论:尽管固相肽合成法是研究Jug r 2线性表位的重要方法,但是由于合成成本高且盲目性大,因此寻找相关辅助方法以提高Jug r 2线性表位的识别效率是重要的研究方向。本研究发现了Jug r 2抗消化肽段具有B细胞线性表位所具备的亲水性和柔韧性的特点,并且通过一级序列和三维定位分析发现了Jug r 2抗消化肽段与预测和真实的B细胞线性表位以及T细胞线性表位重叠。基于Jug r 2抗消化肽段的一级序列和三维定位的分析可能是预测Jug r 2 线性表位的有效策略。
结语:本研究中核桃蛋白所处的体外消化环境较为简单,所采用的体外胃肠模拟消化并不能完全反映核桃蛋白在核桃整个食物基质中的消化行为,特别是由于肠道不同部位发挥作用的消化酶种类的不同,可能导致该研究结果具有一定的局限性。除此之外,核桃主要过敏原Jug r 2抗消化肽是否具有与IgE结合能力仍需更多的核桃过敏患者血清来进一步验证。
原文链接:https://t.cnki.net/kcms/detail?v=z7EBh5rfV004zrz68uaoTRXbEFwYz0qwnlkDT6G4U650DhszJuxQucurWV
_kbH6GWwZnq7XwCQgY9XfNqrvrUWX0xYokWSFWJnklabkw7p_wMvKBDIsSxQ==&uniplatform=NZKPT

